Iberoamérica.
El aprendizaje por refuerzo es un tipo de aprendizaje automático en el que un programa aprende a tomar decisiones probando distintas acciones y recibiendo retroalimentación. Ahora, científicos han logrado aplicar con éxito esta inteligencia artificial a un reto de la biología molecular: el diseño de proteínas.
El equipo investigador, de la Facultad de Medicina de la Universidad de Washington, Estados Unidos, ha desarrollado un nuevo y potente software de diseño de proteínas basado en una estrategia de probada eficacia en juegos de mesa como el ajedrez y el Go, el cual sirvió para fabricar en un experimento posterior cientos de estas sustancias.
«Las proteínas creadas con el nuevo enfoque resultaron más eficaces para generar anticuerpos útiles en ratones», según los autores de la investigación que se publica en la revista Science, quienes sugieren que «este avance podría conducir pronto a vacunas más potentes».
En términos generales, el método podría llevar a una nueva era en el diseño de proteínas, resume un comunicado de la universidad.
«Nuestros resultados demuestran que el aprendizaje por refuerzo puede hacer algo más que dominar juegos de mesa», señala el autor principal David Baker: «si este método se aplica a los problemas de investigación adecuados, podría acelerar el progreso en una variedad de campos».
Las aplicaciones potenciales son enormes, desde el desarrollo de tratamientos más eficaces contra el cáncer hasta la creación de nuevos tejidos biodegradables.
En este aprendizaje por refuerzo un programa aprende a tomar decisiones probando distintas acciones y recibiendo retroalimentación. Y un algoritmo de este tipo puede aprender a jugar al ajedrez, por ejemplo, probando millones de jugadas distintas que conducen a la victoria o la derrota en el tablero. El programa está diseñado para aprender de estas experiencias y mejorar, con el tiempo, en la toma de decisiones.
Para este caso, los científicos dieron al ordenador millones de moléculas sencillas de partida y, a continuación, el programa hizo 10.000 intentos de mejorar aleatoriamente cada una de ellas hacia un objetivo predefinido: el ordenador alargó las proteínas o las dobló de formas específicas hasta que aprendió a darles la forma deseada.
«Nuestro enfoque es único porque utilizamos el aprendizaje por refuerzo para resolver el problema de crear formas de proteínas que encajen como las piezas de un puzzle», afirma Isaac D. Lutz, otro de los firmantes.
«Esto -añade- simplemente no era posible utilizando enfoques anteriores y tiene el potencial de transformar los tipos de moléculas que podemos construir».
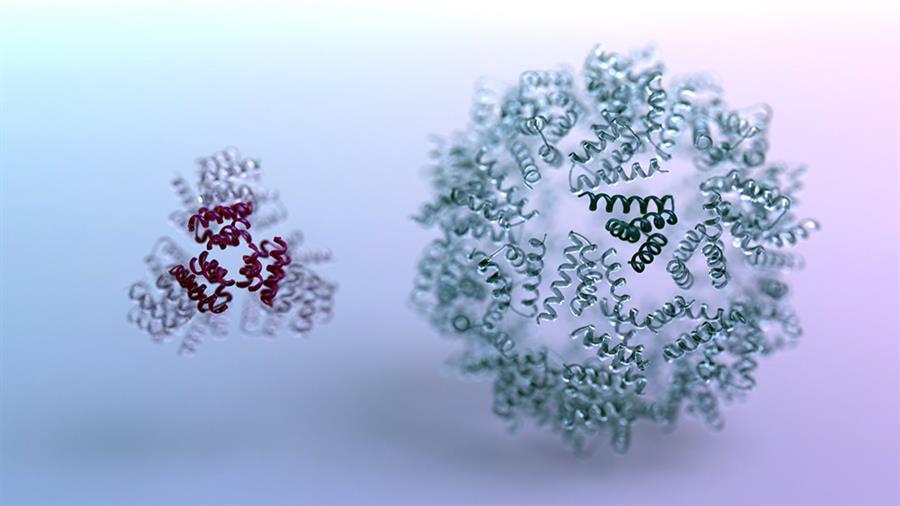
Como parte de este estudio, los científicos fabricaron en el laboratorio cientos de proteínas diseñadas por la inteligencia artificial. Utilizando microscopios electrónicos y otros instrumentos, confirmaron que muchas de las formas de proteínas creadas por el ordenador se hacían realidad en el laboratorio.
«Este método no sólo demostró ser preciso, sino también altamente personalizable. Por ejemplo, le pedimos al programa que creara estructuras esféricas sin agujeros, con agujeros pequeños o con agujeros grandes; su potencial para generar todo tipo de arquitecturas aún está por explorar», detalla Shunzhi Wang.
El equipo se concentró en diseñar nuevas estructuras a escala nanométrica compuestas por muchas moléculas proteicas. Para ello fue necesario diseñar tanto los propios componentes proteicos como las interfaces químicas que permiten el autoensamblaje de las nanoestructuras.
La microscopía electrónica confirmó que numerosas nanoestructuras diseñadas por la IA eran capaces de formarse en el laboratorio.
Para medir la precisión del software de diseño, los científicos observaron muchas nanoestructuras únicas en las que cada átomo estaba en el lugar previsto.
La desviación entre la nanoestructura prevista y la realizada era, por término medio, inferior a la anchura de un solo átomo; es lo que se denomina, apuntan los autores, «diseño atómicamente preciso».
- «Éxito» en pruebas del taxi aéreo en Dubái - julio 1, 2025
- Libro destaca la vida y aporte a la salud de María Isabel Rodríguez - julio 1, 2025
- Netflix retransmitirá en vivo lanzamientos y operaciones espaciales de la NASA - julio 1, 2025